UMAP vs ACP vs Parcoursup
Attention : ce billet est méthodologiquement à la fois expérimental et sans doute discutable. À utiliser donc avec précaution…
Parcoursup ?
Les données concernant l’offre de formations et les voeux effectués dans le cadre de la session 2018 de Parcoursup ont été mises en ligne récemment.
Le jeu de données contient une ligne par formation, avec les caractéristiques de cette formation (établissement, type, filière…) et un certain nombre d’indicateurs sur les voeux effectués pour cette formation et le processus d’admission1. Les données ont été récupérées au format GeoJSON.
On commence par charger les données et calculer un certain nombre d’indicateurs pour chaque formation :
library(tidyverse)
library(sf)
d_orig <- read_sf("data/2019-parcoursup/fr-esr-parcoursup.geojson") %>%
st_drop_geometry()
d <- d_orig %>%
transmute(
# Nombre de places
places = capa_fin,
# Ratio nb de candidats / nb de places
ratio_cand_places = voe_tot / capa_fin,
# Proportion de filles parmi les candidats
cand_f = voe_tot_f / voe_tot,
# Proportion de bac généraux parmi les candidats
cand_bac_gen = nb_voe_pp_bg / nb_voe_pp,
# Proportion de bac techo parmi les candidats
cand_bac_tech = nb_voe_pp_bt / nb_voe_pp,
# Proportion de bac pro parmi les candidats
cand_bac_pro = nb_voe_pp_bp / nb_voe_pp,
# Proportion de boursiers parmi les candidats
cand_bours = (nb_voe_pp_bg_brs + nb_voe_pp_bt_brs + nb_voe_pp_bp_brs) / nb_voe_pp,
# Proportion de candidats classés
classes = nb_cla_pp /nb_voe_pp,
# Proportion de candidats ayant reçu une proposition d'admission
propos = prop_tot / voe_tot,
# Proportion de candidats ayant accepté la proposition d'admission
adm = acc_tot / voe_tot,
# Proportion de filles parmi les admis
adm_f = acc_tot_f / acc_tot,
# Proportion de boursiers parmi les admis
adm_bours = acc_brs / acc_tot,
# Proportion de bac généraux parmi les admis
adm_bac_gen = acc_bg / acc_tot,
# Proportion de bac pro parmi les admis
adm_bac_pro = acc_bp / acc_tot,
# Proportion de bac techno parmi les admis
adm_bac_tech = acc_bt / acc_tot,
# Proportion de bac sans mention parmi les admis
adm_mention_sans = acc_sansmention / acc_tot,
# Proportion de bac mention AB parmi les admis
adm_mention_ab = acc_ab / acc_tot,
# Proportion de bac mention B parmi les admis
adm_mention_b = acc_b / acc_tot,
# Proportion de bac mention TB parmi les admis
adm_mention_tb = acc_tb / acc_tot,
# Proportion d'élèves de la même académie parmi les admis
adm_mm_acad = acc_aca_orig / acc_tot,
)
## Remplacement des NaN générés en cas de division par 0 par des 0
nan_to_zero <- function(v) {
v[is.nan(v)] <- 0
v[is.infinite(v)] <- 0
v
}
d <- d %>%
mutate_all(nan_to_zero)
## On élimine les formations pour lesquelles des proportions sont
## supérieures à 1
invalid <- d %>%
select(-places, -ratio_cand_places) %>%
mutate_all(~{.x > 1}) %>%
rowSums()
d <- d %>% filter(invalid == 0)
d_orig <- d_orig %>% filter(invalid == 0)On a donc un tableau de 10689 formations, et pour chacune d’entre elles 20 variables numériques indiquant le nombre de places ouvertes, le ratio nombre de candidats / nombre de places, la proportion de filles, de type de Bac et de boursiers parmi les candidats et les admis, la proportion de candidats à qui on a proposé une place, etc.
ACP
Quand on a ce type de jeu de données, le réflexe est souvent (en tous cas c’est le mien) d’utiliser une Analyse en Composantes Principales (ACP) pour dégager les axes principaux du nuage de points et visualiser ce nuage en réduisant le nombre de dimensions de 20 à 2 ou 3.
Lançons donc une ACP et affichons l’histogramme des valeurs propres.
library(FactoMineR)
library(explor)
acp <- PCA(d, scale.unit = TRUE, graph = FALSE)
res <- prepare_results(acp)
ggplot(res$eig) +
geom_col(aes(x = factor(dim), y = percent)) +
xlab("Axe") +
ylab("Pourcentage de variance")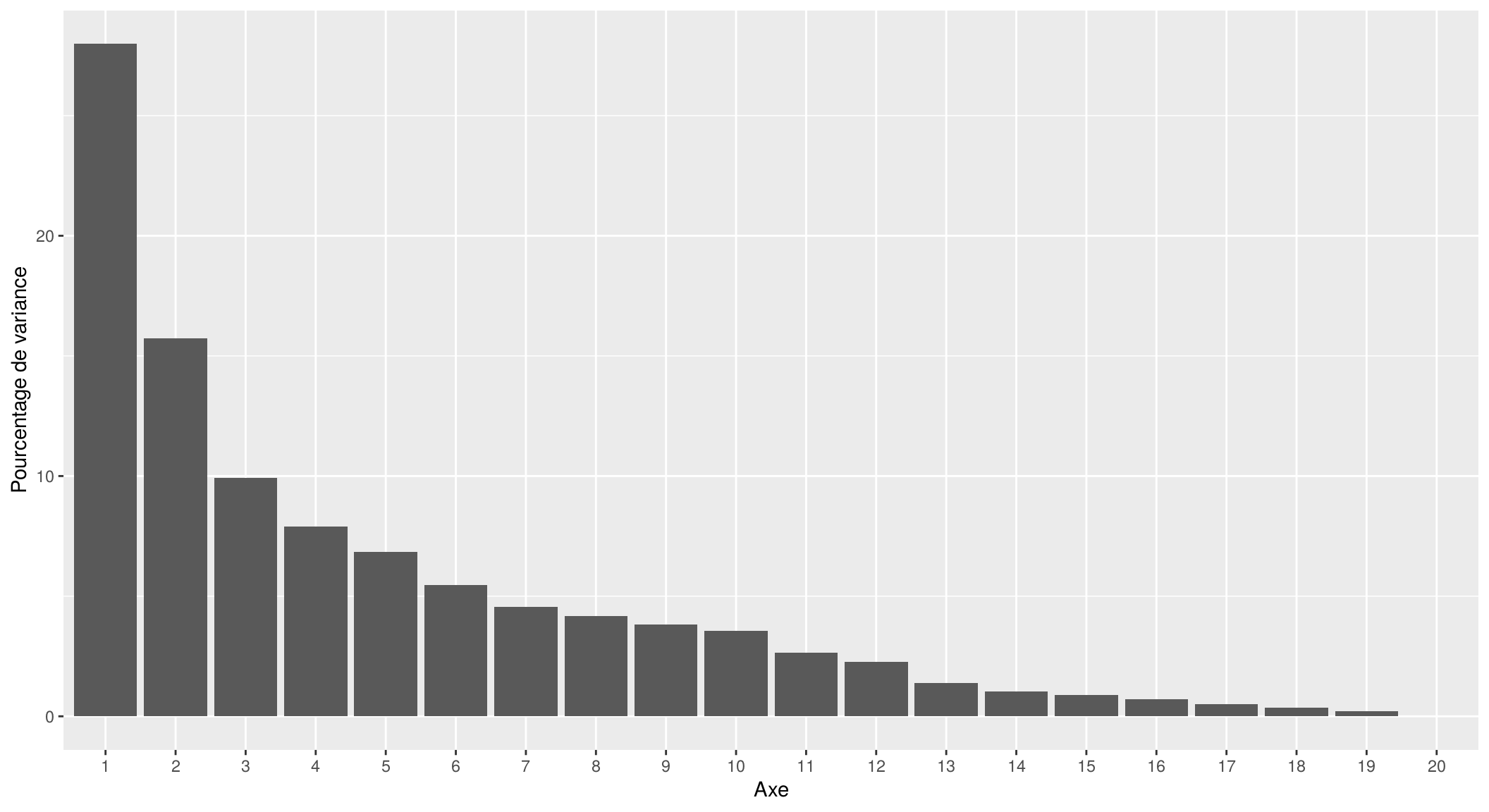
On retrouve deux premiers axes qui se détachent des autres, on peut donc représenter graphiquement l’espace des variables sur ces deux axes :
explor::PCA_var_plot(res, xax = 1, yax = 2, scale_unit = TRUE, labels_position = "auto")L’axe horizontal semble séparer les formations recrutant des bacheliers généraux (à gauche) de celles recrutant plutôt parmi les bacs professionnels ou technologiques (à droite). L’axe vertical, lui, distingue en haut les formations peu sélectives (la plupart des candidats sont classés et admis) et en bas celles qui le sont beaucoup plus (avec des proportions plus élevées de bacheliers avec mention, et un ratio nombre de candidats / nombre de places plus important).
On peut également représenter l’espace des individus :
coords <- tibble(
x = acp$ind$coord[,1],
y = acp$ind$coord[,2]
)
ggplot(coords, aes(x = x, y = y)) +
geom_point(color = "darkblue", alpha = 0.1) +
coord_fixed() +
xlab("Axis 1") +
ylab("Axis 2") 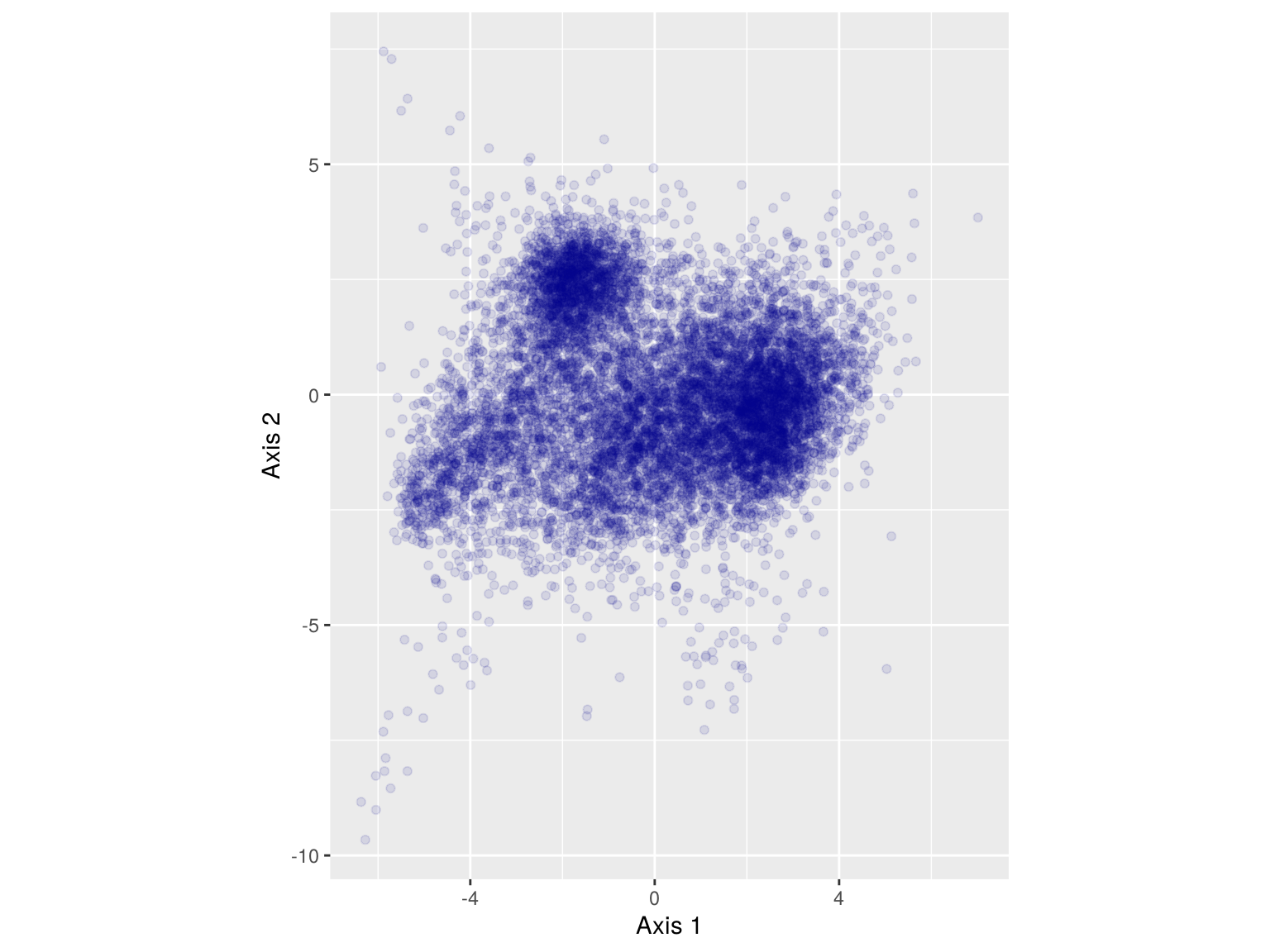
Et on peut colorer cet espace des individus en fonction du type de filière :
coords <- bind_cols(coords, fili = d_orig$fili)
ggplot(coords, aes(x = x, y = y, color = fili)) +
geom_point(alpha = 0.3) +
coord_fixed() +
xlab("Axis 1") +
ylab("Axis 2") +
ggsci::scale_color_d3(name = "Filière", palette = "category10") +
stat_ellipse()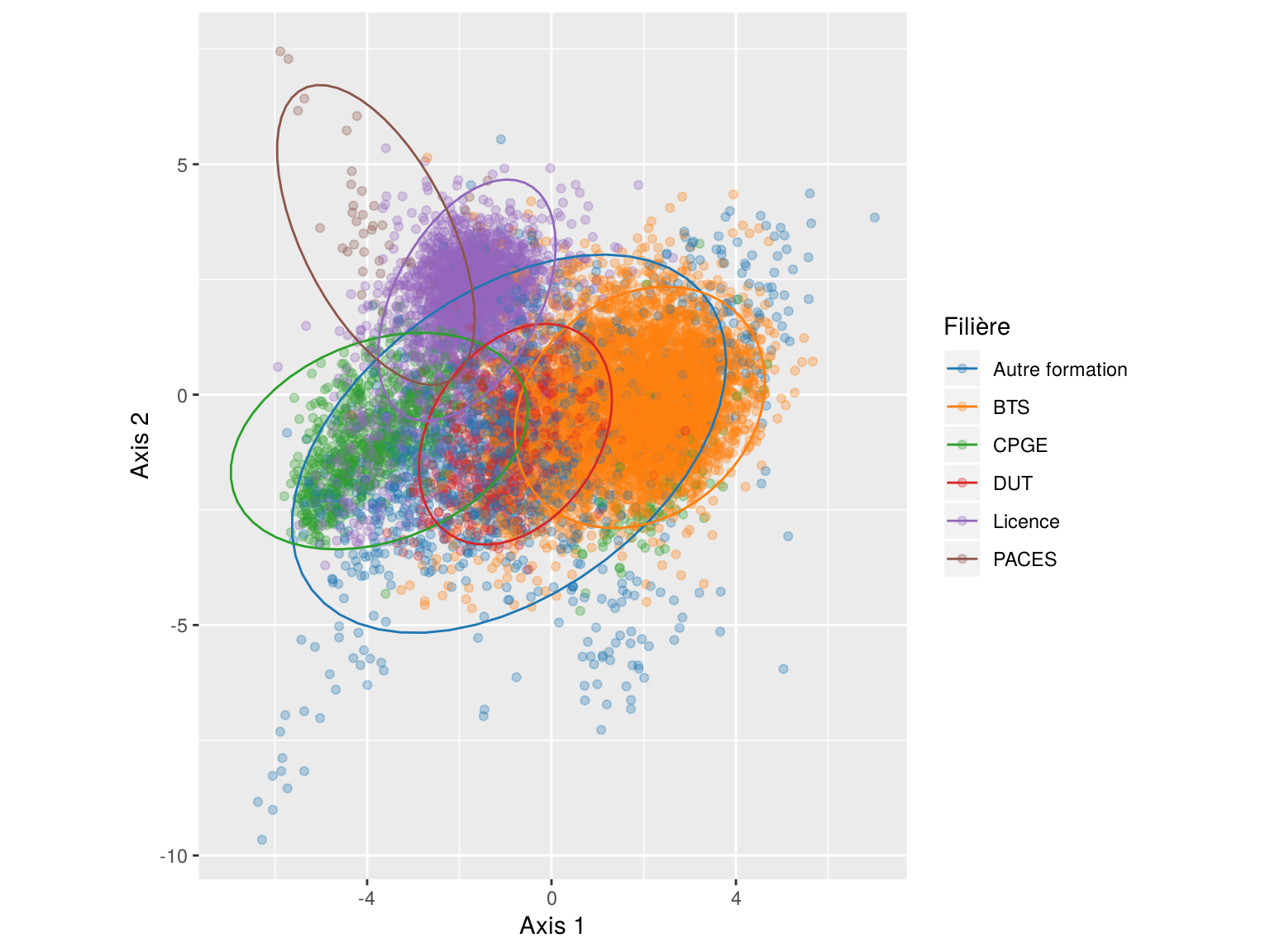
On aperçoit alors une séparation assez forte des différentes filières, (exceptées les Autre formation, mais comme on pouvait s’y attendre).
UMAP
Présentation rapide (et peu claire)
La méthode UMAP (Uniform Manifold Approximation and Projection for Dimension Reduction) est une autre méthode de réduction de dimensionnalité assez récente, puisque l’article qui la présente date de 2018. Elle est assez proche d’une autre famille de méthodes nommée t-SNE mais présente certains avantages par rapport à ces dernières (notamment en termes de rapidité et de préservation de la position globale des groupes dans le résultat final).
La méthode mobilise des notions mathématiques assez complexes, notamment d’analyse topologique, en tous cas nettement trop complexes pour moi… Cependant, en espérant ne pas trop dire de bêtises, le principe global me semble être le suivant :
- pour chaque point, on considère un nombre fixé à l’avance de ses voisins les plus proches. Le rayon de la sphère contenant ces n voisins est utilisé pour définir la base d’une “distance locale” pour ce point.
- cette “distance locale” est utilisée pour attribuer un “poids” (une probabilité) au lien entre le point considéré et chacun de ses n voisins les plus proches. L’algorithme part du principe qu’un point ne peut être totalement isolé, on a donc une probabilité certaine de lien entre un point donné et son plus proche voisin.
- ces ensembles de poids pour chaque point sont utilisés pour calculer un graphe pondéré : chaque noeud est un point, chaque arête est un lien dont le poids est une “probabilité” de lien.
- on applique enfin à ce graphe pondéré un algorithme de force-based layout pour en trouver la meilleure représentation possible en 2 dimensions ou plus.
De nombreuses subtilités reposent notamment dans la manière dont chaque arête est pondérée en fonction de la distance entre les points, et de la manière dont les différentes distances sont “réconciliées” pour obtenir la pondération finale. Pour des informations plus détaillées, on pourra notamment se référer à la page d’introduction How UMAP works.
Application à Parcoursup
On va appliquer UMAP à notre jeu de données grâce au package R uwot. On utilise l’argument
scale = "scale" pour centrer-réduire les variables avant calcul :
library(uwot)
um <- umap(d, n_neighbors = 15, min_dist = 0.01, scale = "scale")Le temps de calcul n’est que de quelques secondes, un peu plus long que celui de l’ACP mais tout à fait raisonnable. Le résultat est une simple matrice avec deux colonnes indiquant les coordonnées de chaque point dans les deux dimensions.
On peut à partir de cette matrice représenter la position de chaque point dans ce nouvel espace réduit, comme pour une ACP :
res_um <- tibble(x = um[,1], y = um[,2])
ggplot(res_um, aes(x = x, y = y)) +
geom_point(color = "darkblue", alpha = 0.1) +
coord_fixed()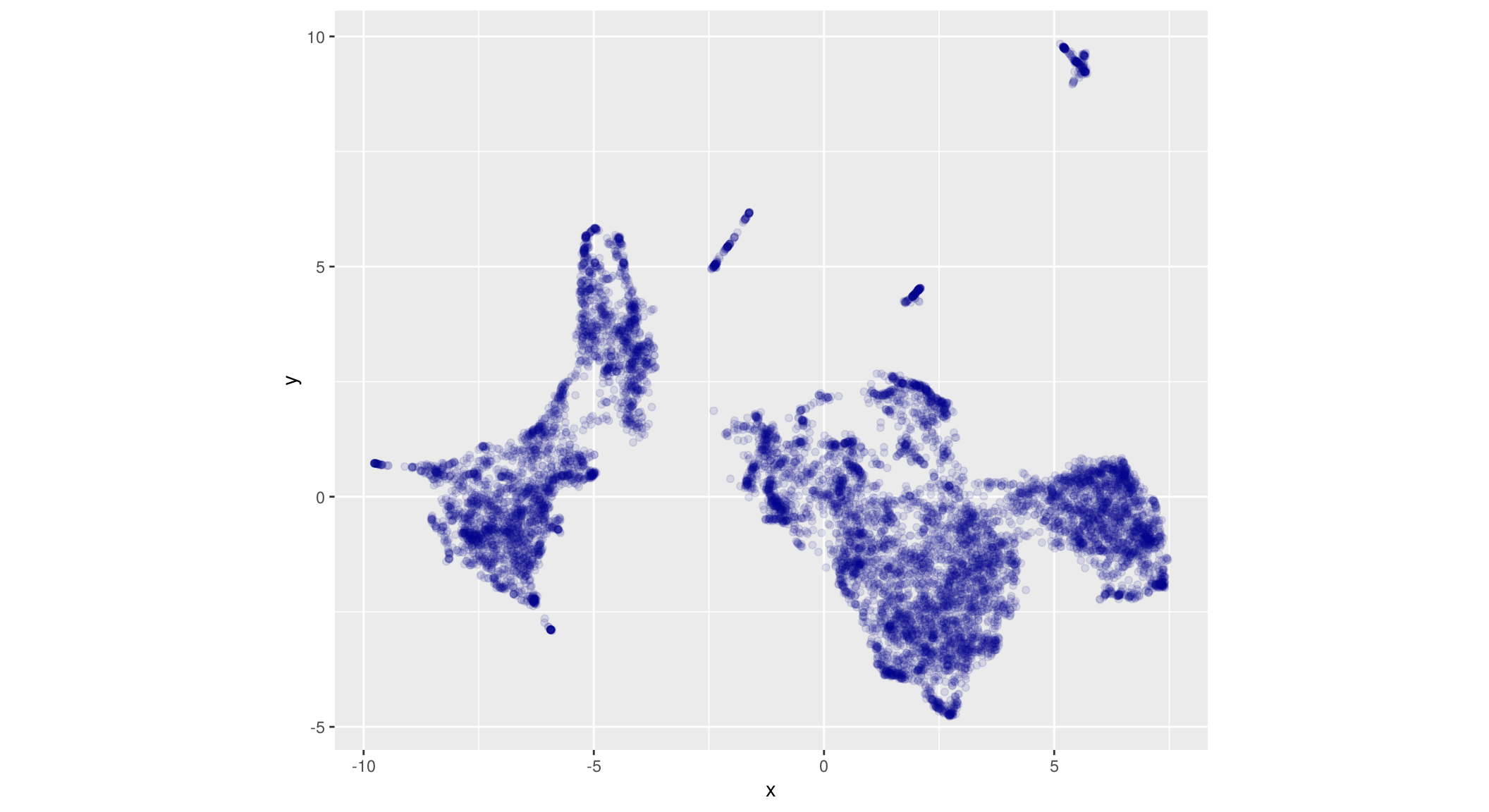
On constate d’emblée que le résultat est visuellement très différent de celui de l’ACP : des grands groupes se distinguent beaucoup plus nettement, et à l’intérieur de ces groupes on peut repérer des “amas” plus petits et assez nombreux.
Une grosse différence avec l’ACP est qu’ici les axes ne sont pas interprétables, ils n’ont aucun sens particulier : UMAP se rapproche davantage d’une “carte”, où longitude et latitude n’ont pas réellement d’intérêt en soi, et où c’est la position des éléments les uns par rapport aux autres qui prime.
Paramètres de la méthode
La fonction umap accepte de nombreux arguments, dont certains sont des paramètres
importants qui peuvent assez considérablement modifier le résultat obtenu. Parmi ceux-ci on notera
surtout :
n_neighbors: le nombre de plus proches voisins considérés dans le calcul du graphe de proximités. C’est le paramètre le plus important : avec une valeur élevée, le résultat donnera une vue plus globale de l’ensemble de points ; avec une valeur plus faible il mettra plus en valeur des particularités locales. Cette valeur est en général comprise entre 2 et 100 (15 par défaut).min_dist: c’est la distance minimale entre deux points dans l’affichage final. Une valeur basse donnera des points beaucoup plus regroupés, une valeur élevée des points plus répartis (0.01 par défaut).n_components: le nombre de dimensions du résultat final (2 par défaut).metric: le type de distance utilisée. Peut valoir"euclidean"(valeur par défaut), mais aussi"cosine","manhattan","hamming"et"categorical". Il est également possible de spécifier des distances différentes selon les variables.
Contrairement à l’ACP, ces paramètres ont une grande influence sur les résultats. Pour l’illustrer on
relance UMAP sur notre jeu de données avec différentes valeurs de n_neighbors et
min_dist :
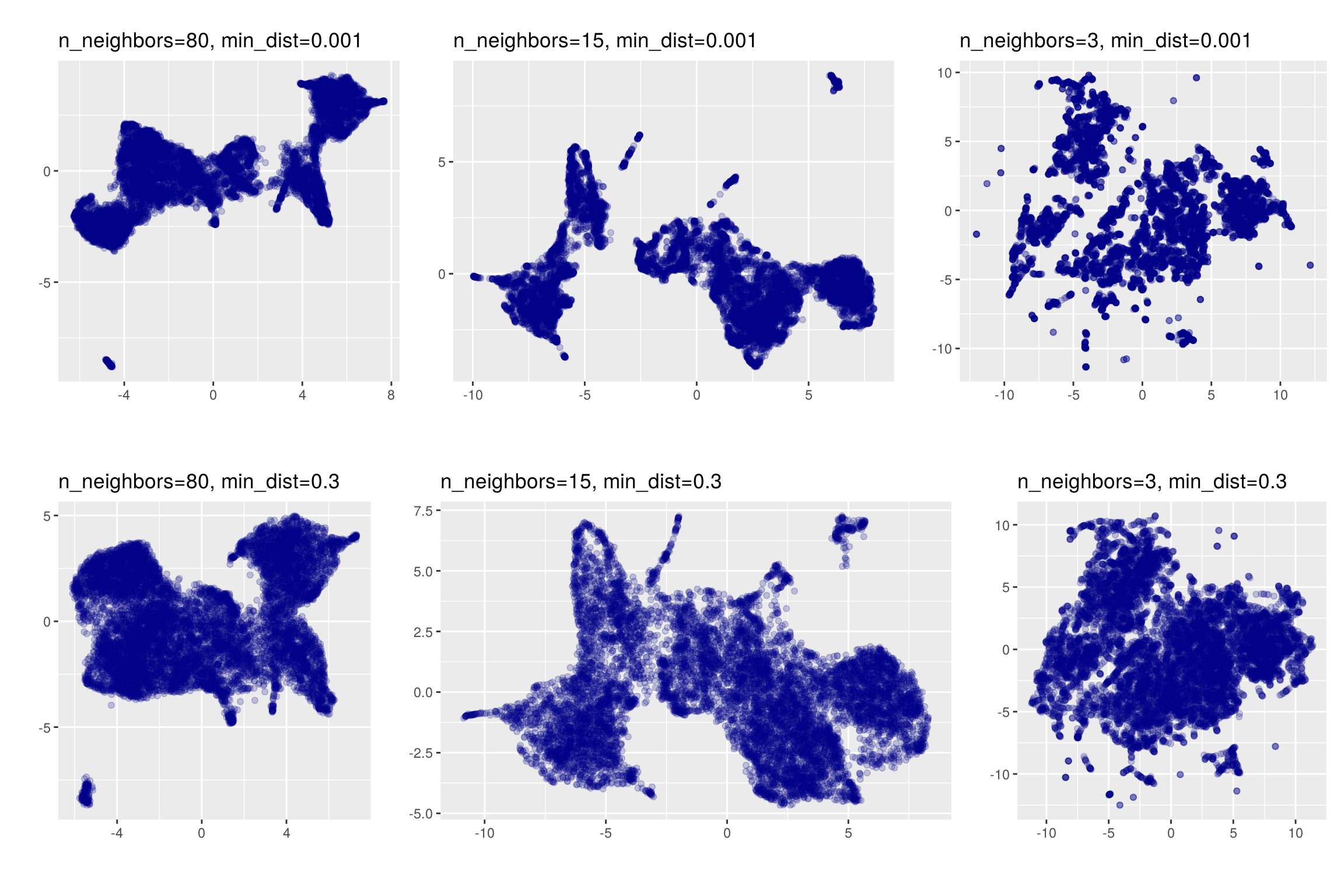
On obtient des résultats visuellement très différents. Quand on augmente la valeur de
min_dist (graphiques de la ligne du haut vs ceux de la ligne du bas), les points
sont moins regroupés mais certaines structures sont moins visibles. Quand on diminue la valeur de
n_neighbors, on voit moins bien la structure globale au profit des structures locales.
Il est donc très important de lancer UMAP avec différents paramètres et de comparer les résultats obtenus.
UMAP est non déterministe
Une autre particularité potentiellement déroutante d’UMAP est qu’il s’agit d’une méthode non déterministe : appliquée deux fois de suite sur les mêmes données avec les mêmes paramètres, elle ne donnera pas exactement les mêmes résultats.
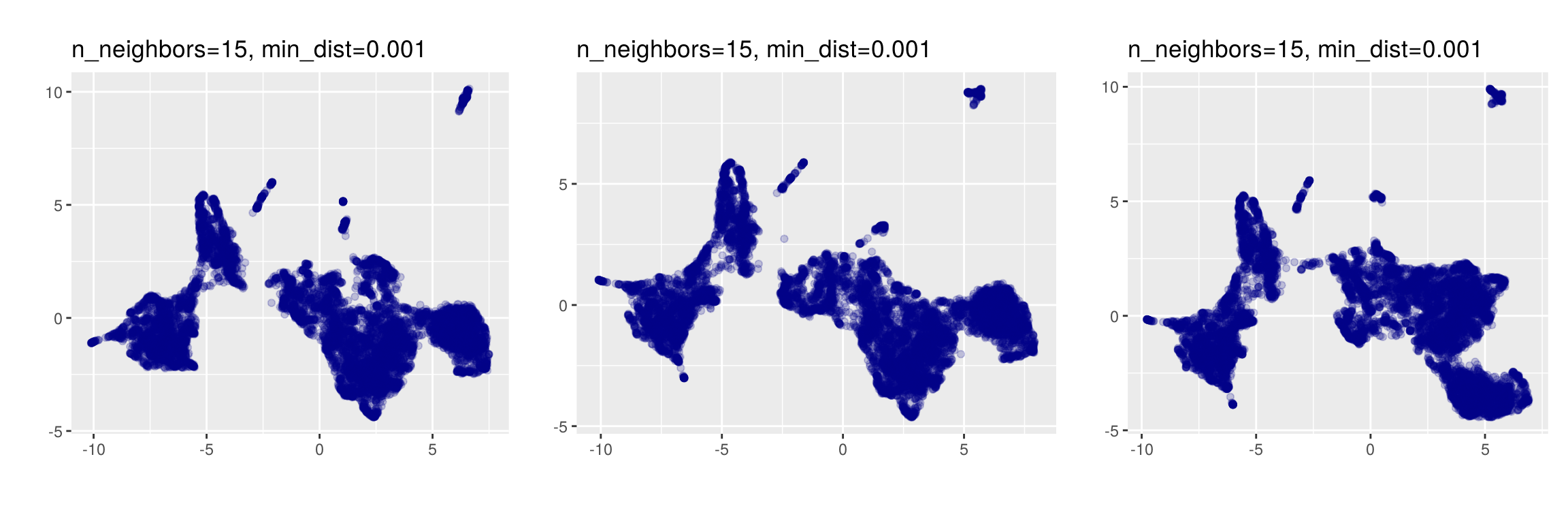
Si les résultats ne sont pas radicalement différents, la position ou la forme de certains groupes de points peut varier. Il est donc là aussi souhaitable de lancer plusieurs fois la méthode avec les mêmes paramètres pour évaluer la variabilité des résultats.
Si on souhaite pouvoir reproduire une analyse à l’identique, on peut initialiser le générateur de
nombre aléatoire avec set.seed juste avant d’appeler umap :
set.seed(1515)
um <- umap(d, n_neighbors = 15, min_dist = 0.01, scale = "scale")Comparaison des deux méthodes
Dans ce qui suit on lance UMAP sur nos données avec une valeur de n_neigbors à 30 :
set.seed(1337)
um <- umap(d, n_neighbors = 30, min_dist = 0.01, scale = "scale")Et on compare la répartition des individus entre cette UMAP et l’ACP précédente :
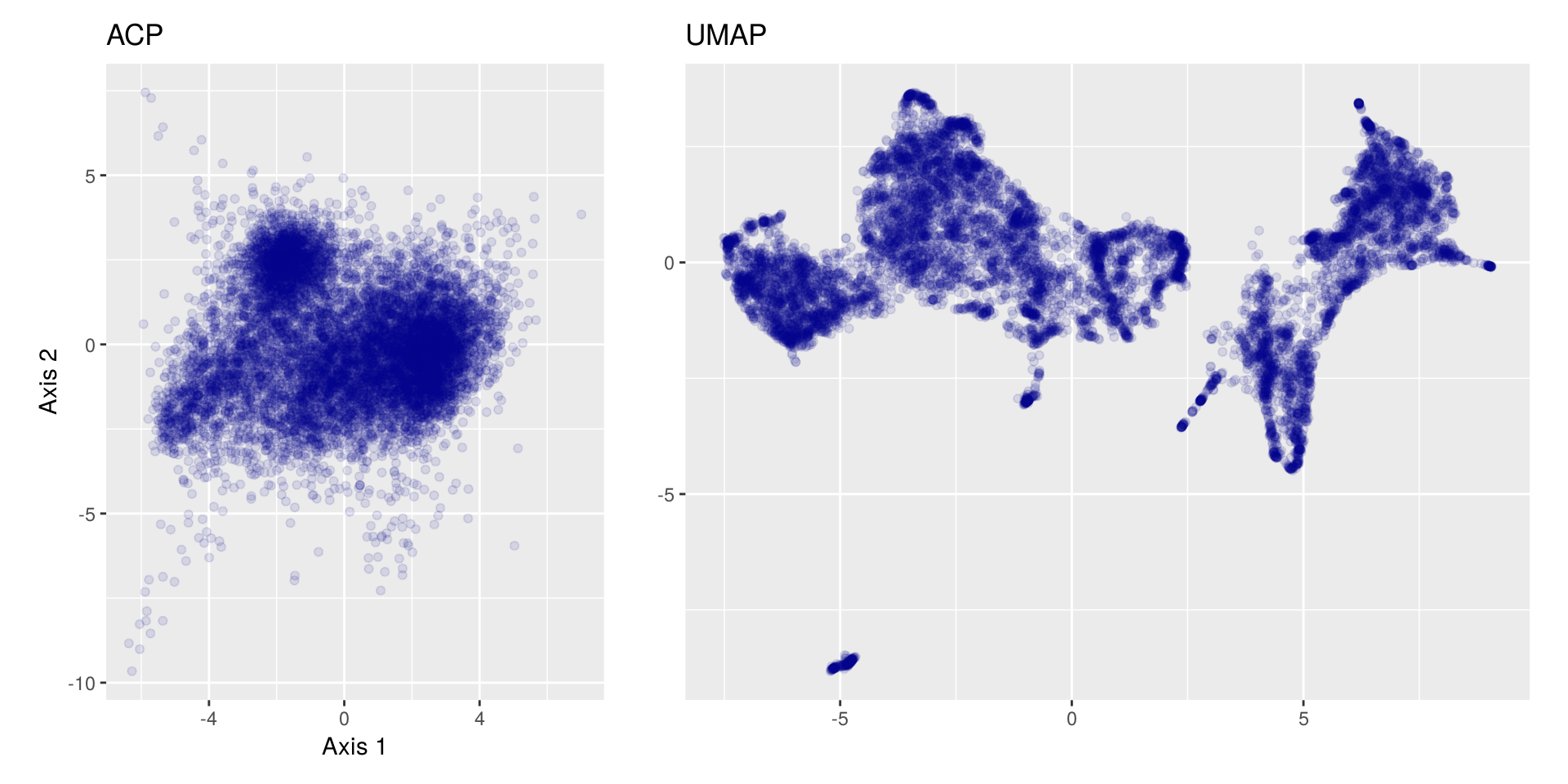
Comme on l’a déjà souligné, des groupes se détachent beaucoup plus nettement dans le résultat d’UMAP, que ce soit globalement ou localement.
On colore les points dans les deux cas en fonction du type de filière :
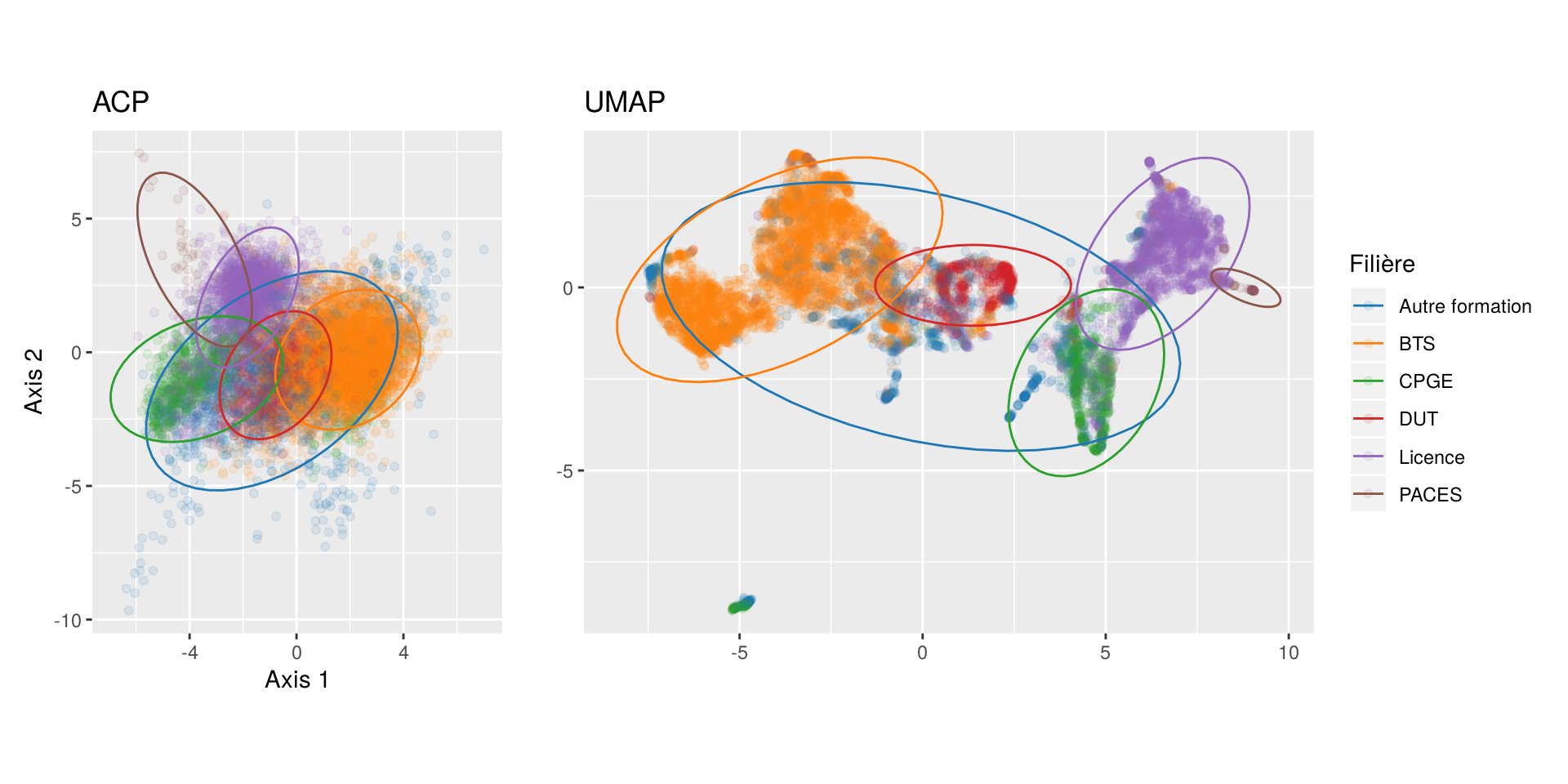
Si l’ACP parvient à séparer visuellement assez bien les différentes filières de formation, dans le cas d’UMAP la séparation est flagrante. On arrive globalement à isoler les filières de manière très nette, seulement à partir des 20 indicateurs numériques utilisés.
On peut aussi colorer les observations en fonction des valeurs de ces indicateurs. On commence par la proportion de filles parmi les candidats admis :
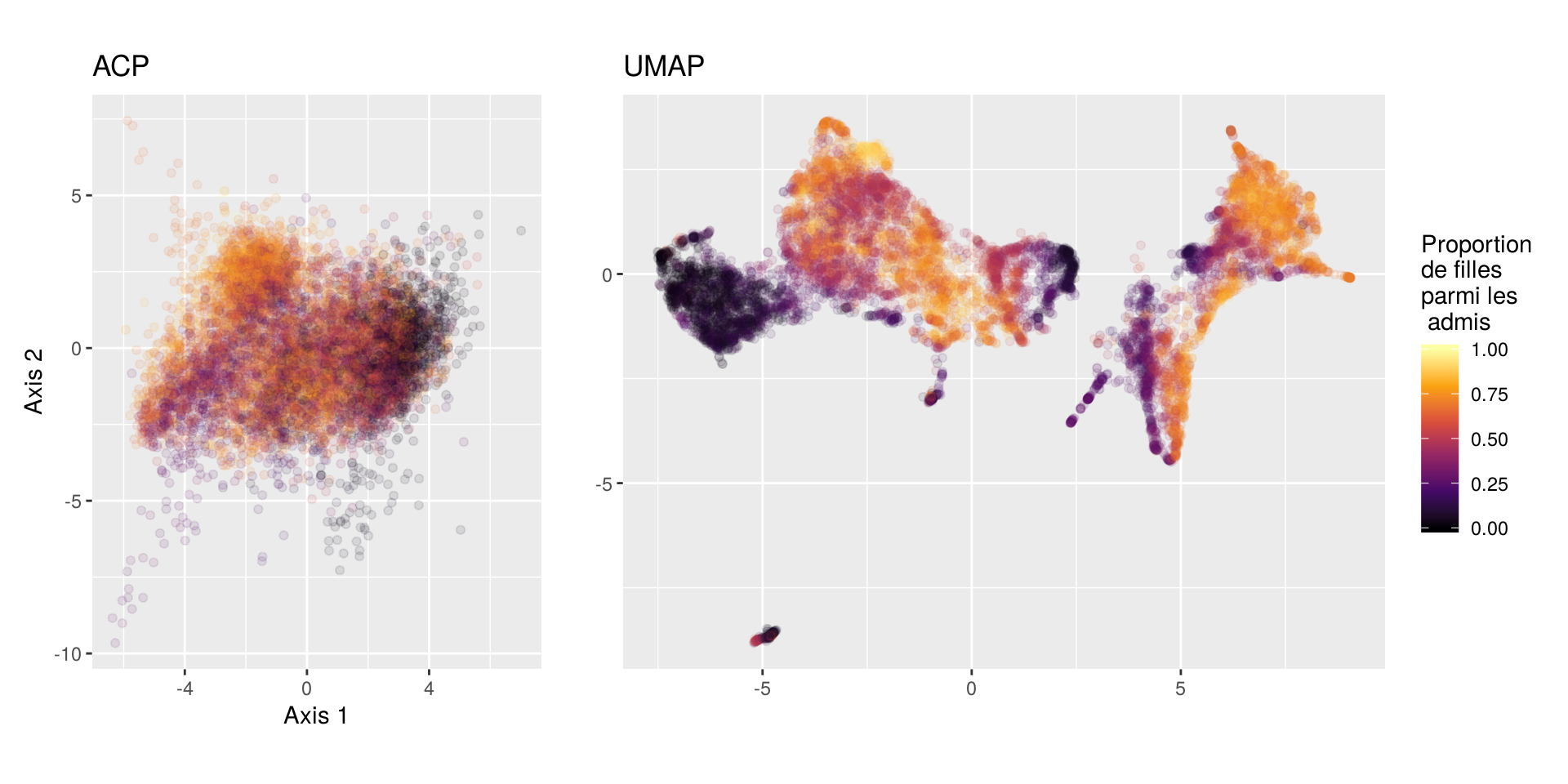
Là où l’ACP montre des différences au niveau global (avec notamment une proportion de filles moindre à la droite du nuage), les distinctions sont beaucoup plus fines avec UMAP. On note des écarts importants à l’intérieur du groupe des BTS (avec notamment un sous-groupe constitué quasi uniquement de garçons), mais aussi dans les autres groupes comme DUT, CPGE ou même licences, ce que l’ACP ne fait pas apparaître aussi nettement.
On colore maintenant les points en fonction de la proportion de bac pro parmi les candidats admis :
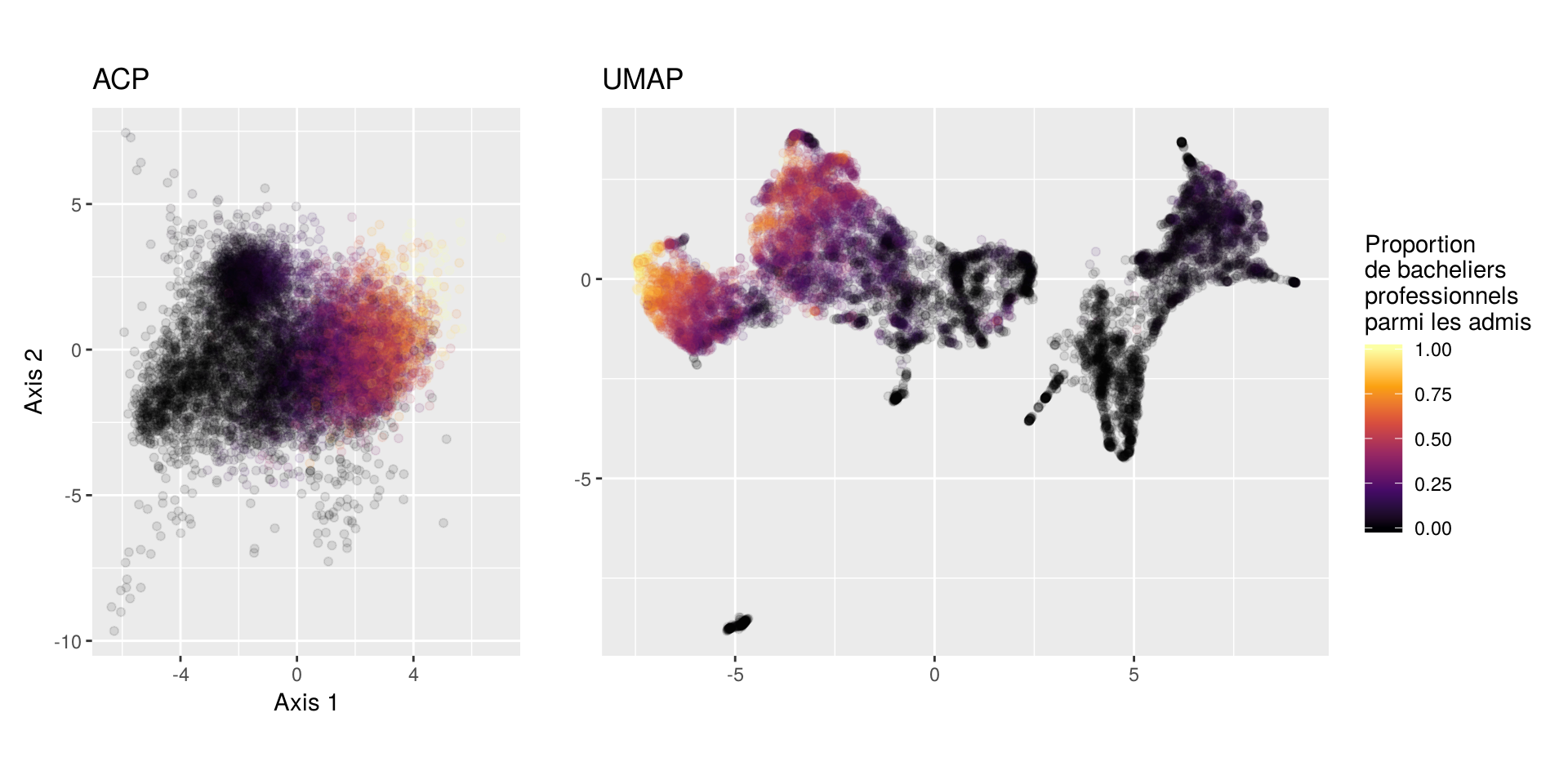
Ici la différence entre ACP et UMAP est moins flagrante : on retrouve globalement un gradient assez régulier de la proportion de bacheliers professionnels.
On continue avec la proportion de bacheliers technologiques :
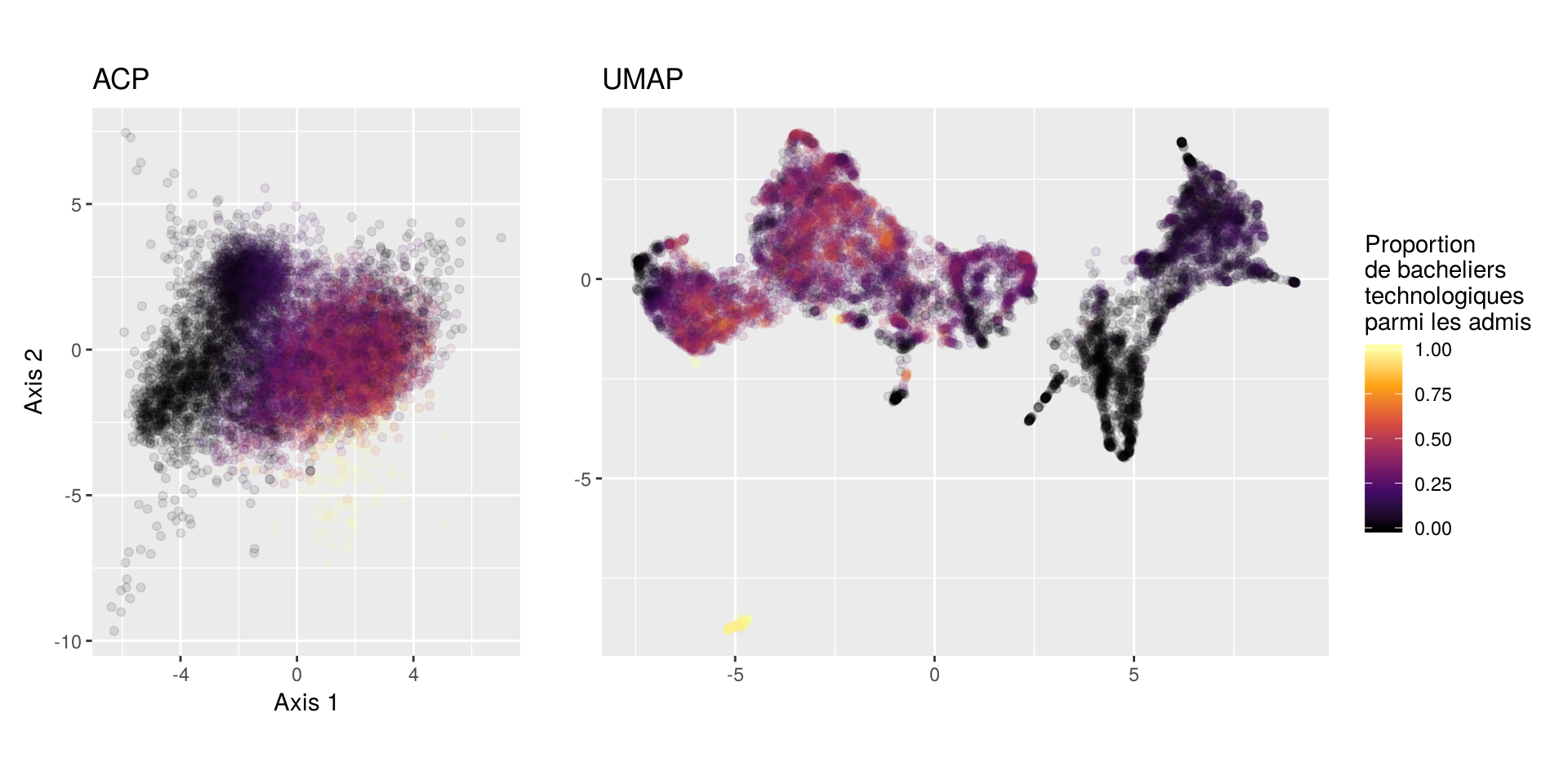
Là où l’ACP montre un gradient plutôt progressif, UMAP permet de voir des variations plus locales, notamment à l’intérieur du groupe de BTS. Mais surtout, on repère un petit groupe de formations isolées qui se caractérisent par une très forte proportion de bacheliers technologiques parmi les admis. Il s’agit donc sans doute de formations avec un profil assez spécifique.
Le graphique suivant montre la proportion de boursiers parmi les candidats :
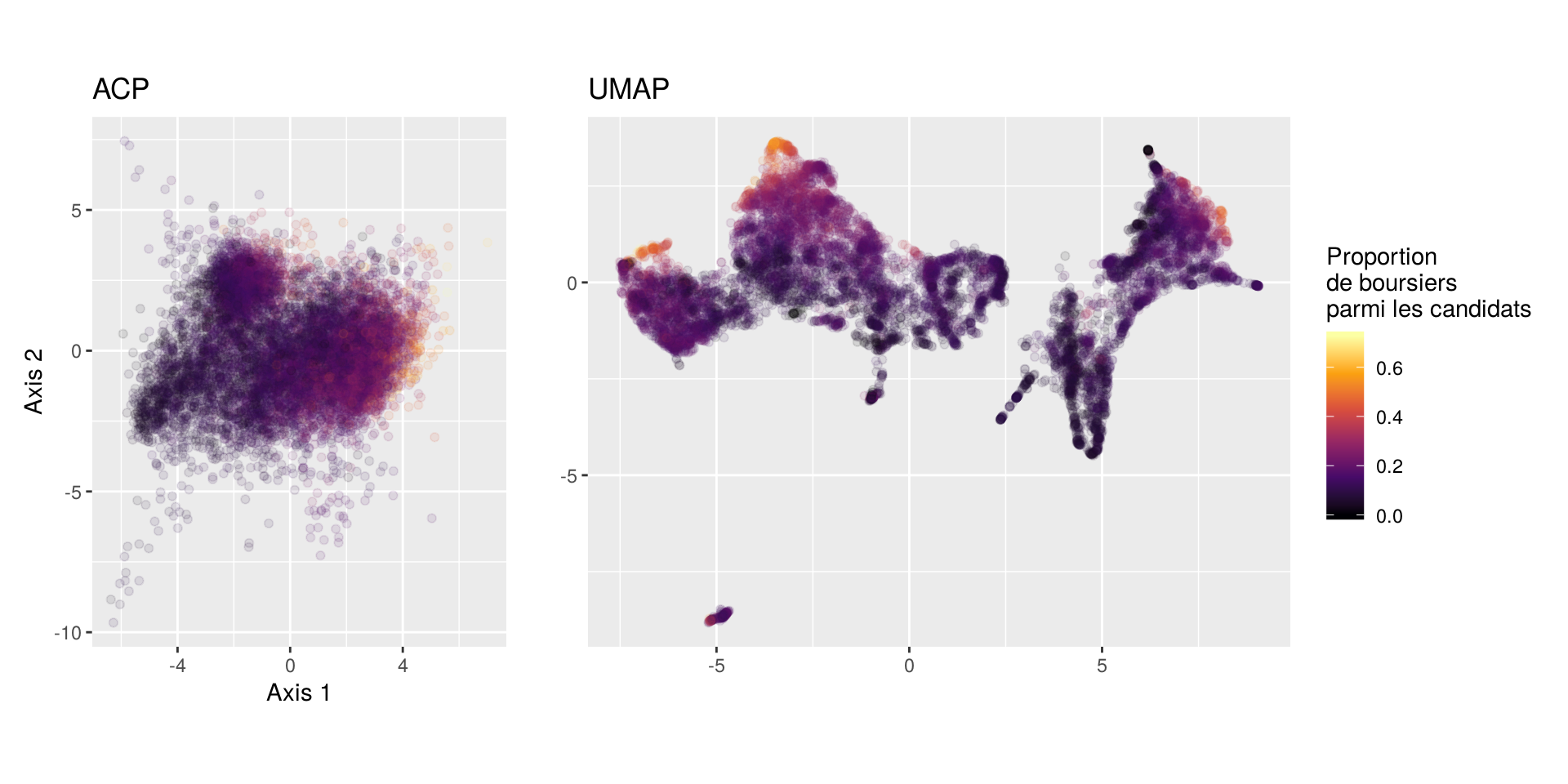
Là où l’ACP semble permettre de distinguer deux zones avec des proportions de boursiers plus importantes, UMAP en indique assez nettement au moins trois, deux dans le groupe des BTS et une dans le groupe des licences.
Enfin, on termine avec la proportion de bacheliers ayant eu une mention très bien parmi les admis :
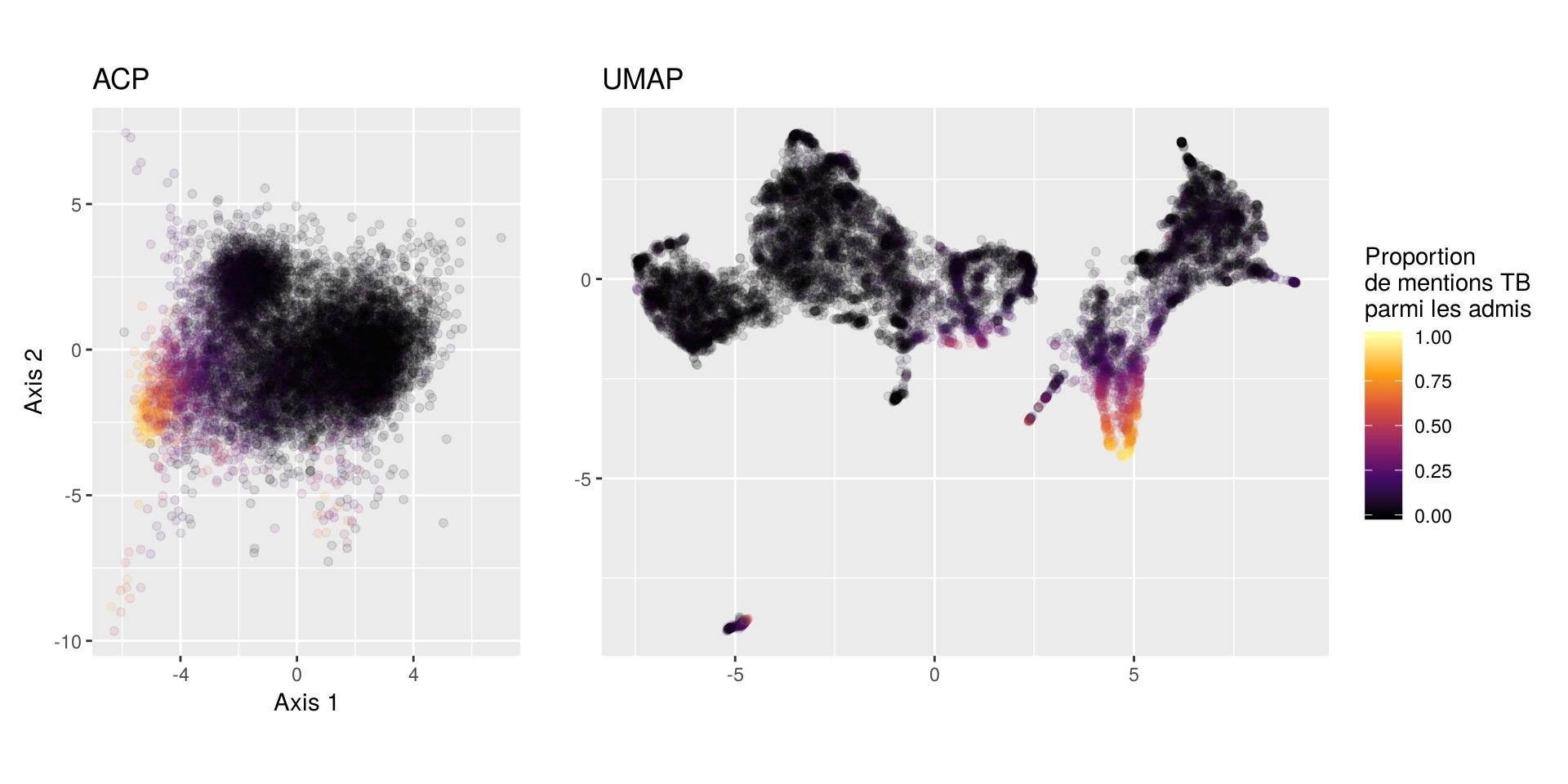
Si les deux méthodes permettent de mettre en évidence des formations avec une forte proportion de mentions très bien dans le groupe des CPGE, la distinction semble plus nette avec UMAP, et celle-ci permet également repérer une zone avec des proportions plutôt élevées mais cette fois dans le groupe des BTS et des DUT, ce que l’ACP ne permet pas de voir.
Conclusion
Comparaison
Si on devait comparer les deux méthodes sur cet exemple rapide, on pourrait souligner que l’ACP a plusieurs avantages :
- le calcul est très rapide, même pour des tableaux de grande dimension
- la méthode est déterministe, on obtient toujours les mêmes résultats à partir des mêmes données
- les axes résultant du calcul sont “interprétables”, on peut leur donner du sens
- le principe de la méthode reste relativement intuitif si on le voit comme “la meilleure rotation possible du nuage de points”
De son côté, UMAP semble avoir des avantages dans la finesse des résultats obtenus : il semble possible de distinguer plus de structures, à la fois locales et globales, et de mettre en évidence davantage de différences dans la répartition des valeurs des indicateurs numériques utilisés pour le calcul.
Limites
Évidemment, cette comparaison rapide a de nombreuses limites, dont la première est la question de sa généralisation : UMAP fonctionne bien sur ce jeu de données, mais rien ne nous dit que ce sera le cas sur un autre. En particulier, on sait qu’UMAP, du fait d’approximations visant à accélérer les calculs, fonctionne moins bien sur de petites populations. Elle est également moins intéressante sur des populations pour lesquelles la structure globale est plus importante que les structures locales.
L’aspect non déterministe et l’importance des paramètres dans les résultats obtenus peut aussi paraître rédhibitoire pour certaines personnes ou certaines applications. Enfin, un des risques d’UMAP est de faire apparaître des structures au niveau local là où il n’y aurait que du bruit : il peut donc être risqué de l’appliquer aveuglément, notamment sur des données pour lesquelles on ne pourrait pas contrôler les résultats obtenus a posteriori 2.
Et ensuite ?
Quand on fait une ACP, il est fréquent d’enchaîner avec une méthode de classification, notamment de type CAH, pour mettre formellement en évidence des groupes d’observation.
Il est donc tout naturel de vouloir faire de même après l’utilisation d’UMAP. Cependant, les résultats de celle-ci ne respectant pas les distances entre les points, seulement leur répartition, il est déconseillé d’utiliser les méthodes traditionnelles du type CAH ou k-means.
Des alternatives existent cependant, et c’est justement l’objet du prochain billet (et peut-être même d’un package R).
-
Pour plus d’informations, se référer à la documentation associée.↩
-
Pour une liste plus complète des limites inhérentes à UMAP, on pourra se reporter à la section Weaknesses de l’article original↩